31. Reducción de dimensionalidad con PCA#
Principal Component Analysis (PCA) es un método clásico que permite proyectar nuestros datos y reducir su dimensionalidad. Primero se revisa el método de forma teórica y luego su implementación en la librería sklearn.
31.1. Formalismo matemático#
PCA es un procedimiento estadístico que busca una transformación ortogonal que maximice la varianza de los datos.
Matemáticamente, podemos escribir un conjunto de datos \(\{x_i\}\) con \(i=1,2,\ldots, N\) y \(x_i \in \mathbb{R}^D\), como una matriz \(X \in \mathbb{R}^{N\times D}\). La transformación en PCA está representada por una matriz \(W \in \mathbb{R}^{D\times D}\) y los datos transformados se obtienen mediante una multiplicación matricial
Una transformación es ortogonal si cumple \(W^T W = I\) es decir que la transformación traspuesta es equivalente a su inversa. Para encontrar una transformación que maximice la varianza de los datos debemos obtener primero \(C\) la matriz de covarianza de \(X\).
Asumiendo que las columnas de \(X\) están centradas (su media es cero), la covarianza de \(X\) se define como
donde \(C \in \mathbb{R}^{D\times D}\).
Luego, la matriz de covarianza de los datos transformados \(\hat X\) es
Con lo anterior podemos escribir la siguiente función objetivo
Utilizando multiplicadores de Lagrange para incluir la restricción en el objetivo y derivando e igualando a cero se obtiene lo siguiente
donde \(\Lambda = \lambda I\) y \(\lambda = (\lambda_1, \lambda_2, \ldots, \lambda_D)\).
Importante
Este problema de optimización corresponde al problema de los valores y vectores propios (\(\lambda\) y \(W\)) de la matriz \(C\).
31.2. Implementación utilizando scipy#
A modo de ejemplo sean los siguientes datos bidimensionales
import numpy as np
X = np.random.multivariate_normal([0, 0], [[0.5, -0.7], [-0.7, 1]], size=1000)
Utilicemos PCA para encontrar los ejes coordenados de máxima varianza y graficarlos. Para resolver el problema de valores propios podemos utilizar scipy.linalg.eigh:
import scipy.linalg
# Restamos la media
X_ = X - np.mean(X, axis=0, keepdims=True)
# Calculamos la covarianza
C = np.dot(X_.T, X_)/len(X_)
# Calculamos los valores y vectores propios de la covarianza
L, W = scipy.linalg.eigh(C)
# Proyectamos
U = np.dot(X, W)
El resultado gráfico es:
%matplotlib inline
import matplotlib.pyplot as plt
arrow_args = {'width': 0.05, 'length_includes_head': True, 'alpha': 0.5}
fig, ax = plt.subplots(1, 2, figsize=(8, 4), tight_layout=True,
sharex=True, sharey=True)
ax[0].scatter(X[:, 0], X[:, 1], s=10);
for i, c in enumerate(['g', 'r']):
ax[0].arrow(0, 0, W[i, 0], W[i, 1], color=c, **arrow_args)
ax[0].set_aspect('equal');
ax[1].set_aspect('equal');
ax[0].set_xlim([-3.5, 3.5])
ax[1].scatter(U[:, 0], U[:, 1])
ax[1].spines['bottom'].set_color('g')
ax[0].set_ylabel('Atributo 1')
ax[0].set_xlabel('Atributo 2')
ax[1].set_ylabel('Componente principal 1')
ax[1].set_xlabel('Componente principal 2')
ax[1].spines['left'].set_color('r')
ax[0].set_title('Datos originales')
ax[1].set_title('Datos proyectados');
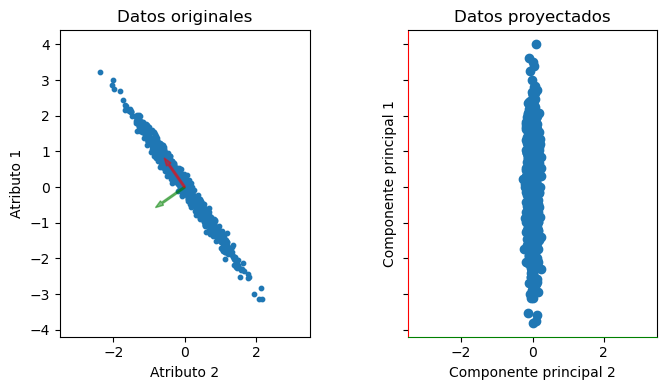
Nota
El eje rojo acumula un 99.5% de la varianza
El eje verde es ortogonal al rojo
Los nuevos ejes están decorrelacionados c/r a los originales
31.3. Reducción de dimensionalidad#
Una aplicación típica de PCA es la reducción de dimensionalidad. Recordemos:
La matriz \(W\) tiene las mismas dimensiones que \(C\)
Las columnas de \(W\) son los vector propios
Cada vector propio tiene un valor propio asociado
Nota
El valor propio \(\lambda_i\) asociado a la columna \(i\) de \(W\) corresponde a la “cantidad de varianza” de dicha columna.
Para obtener una proyección que disminuya la dimensionalidad necesitamos una matriz
con \(\hat D < D\). Podemos obtener \(\widehat W\) uniendo un subconjunto de las columnas de \(W\). En particular nos interesa proyectar a los vectores propios de mayor varianza.
El valor de \(\hat D\) se escoge de acuerdo a la aplicación:
Para tareas de visualización de datos: \(\hat D = 2\) o \(3\)
Para tareas más generales de reducción de dimensionalidad o decorrelación de atributos, un criterio típico es seleccionar \(\hat D\) tal que la suma de varianza acumulada sea al menos un 95%
Importante
Si proyectamos con \(\widehat W\) la transformación no es invertible porque estamos “descartando” información. Es una compresión de tipo lossy (con pérdida).
31.4. Implementación de PCA en sklearn#
PCA está incluido en el módulo sklearn.decomposition. El constructor y sus argumentos más importantes son:
PCA(n_components=None, # Cantidad (int) o porcentaje de varianza (float) a retener
copy=True, # Si es falso, la data transformada reemplaza la original
whiten=False, # Reescala los datos para que tengan igual dispersión
...
)
Los métodos más importantes de la clase PCA son:
fit(X): Calcula la matriz de covarianza y los vectores y valores propiostransform(X): Retorna la proyección de \(X\)fit_transform(X):fitytransformen un solo pasoinverse_transform(hatX): Retorna los datos al espacio original (con pérdidas si \(\hat D < D\))get_covariance(): Retorna la matriz de covarianza
Los atributos más importantes son:
components_: Los componentes principales (vectores propios)singular_values_: Los valores propiosexplained_variance_ratio_: El porcentaje de varianza asociado a cada vector propio
Ejemplo
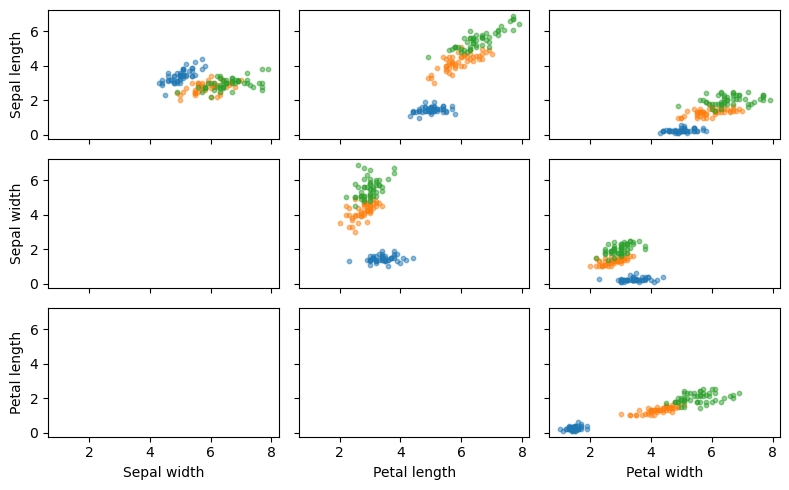
Sea la siguiente base de datos con cuatro atributos numéricos asociados a las características de un conjunto de 150 flores del género Iris separadas en 3 clases.

A continuación se presenta un gráfico de dispersión para visualizar las relaciones entre los atributos
from sklearn.datasets import load_iris
iris_set = load_iris()
X, y, y_names = iris_set['data'], iris_set['target'], iris_set['target_names']
fig, ax = plt.subplots(3, 3, figsize=(8, 5), tight_layout=True, sharex=True, sharey=True)
for i in range(3):
for j in range(i, 3):
for y_ in range(3):
ax[i, j].scatter(X[y==y_, i], X[y==y_, j+1], s=10, alpha=0.5)
ax[0 ,0].set_ylabel('Sepal length')
ax[1 ,0].set_ylabel('Sepal width')
ax[2 ,0].set_ylabel('Petal length')
ax[2 ,0].set_xlabel('Sepal width')
ax[2 ,1].set_xlabel('Petal length')
ax[2 ,2].set_xlabel('Petal width');
Para hacer una visualización más concisa podemos usar PCA para reducir la dimensión de los datos de 4 a 2
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
hatX = pca.fit_transform(X)
Los valores propios:
pca.singular_values_
array([25.09996044, 6.01314738])
El porcentaje de varianza de cada valor propio:
pca.explained_variance_ratio_
array([0.92461872, 0.05306648])
Y finalmente los vectores propios:
hatW = pca.components_
hatW
array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102]])
Podemos estudiar la contribución de cada atributo a los nuevos ejes:
fig, ax = plt.subplots(1, 2, figsize=(8, 4), tight_layout=True)
for y_, name in enumerate(y_names):
ax[0].scatter(hatX[y==y_, 0], hatX[y==y_, 1], s=10, label=name)
ax[0].legend();
for ax_ in ax:
ax_.set_ylabel('Vector propio 2'); ax_.set_xlabel('Vector propio 1')
ax[1].plot([0, 0], [-1 ,1], 'k--', alpha=0.5)
ax[1].plot([-0.5, 2], [0, 0], 'k--', alpha=0.5)
for i, name in enumerate(iris_set.feature_names):
ax[1].arrow(0, 0, hatW[0, i], hatW[1, i], color='b', **arrow_args)
ax[1].text(hatW[0, i], hatW[1, i], name)
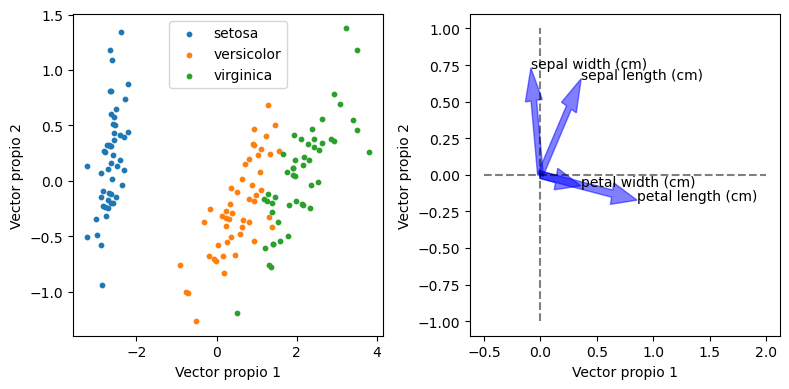
La figura de la izquierda son los datos de iris proyectados en los componentes principales más importantes
La figura de la derecha muestra la contribución de los atributos originales a cada uno de los vectores propios
Nota
El tipo de flor, es decir las clases de Iris, pueden separarse en el eje del vector propio 1. Podemos ver también que las variables que tienen que ver con el sépalo están alineadas con el vector propio 2 (ángulo menor), en cambio las variables que tienen que ver con el pétalo están alineadas con el vector propio 1. En resumen la clase de la flor tiene mayor relación con el pétalo que con el sépalo.