39. Acelerando Python con Cython#
La implementación más utilizada del intérprete de Python está escrita en C y se llama CPython.
Nota
También existen otras implementaciones de Python, como PyPy (incorpora compilación JIT), Jython (escrito en Java) o IronPython.
CPython compila el código escrito en Python en un código de máquina (binario) de forma transparente al usuario. Luego CPython interpreta el binario.
Nota
Si es de vuestro interés, es posible estudiar el código binario usando el módulo dis de Python.
Por lo tanto, si sabemos C, podemos usar la API de Python/C para:
Escribir módulos de C que puedan llamarse desde Python
Hacer interfaces entre código C y Python
Sin embargo, la API es un poco complicada, por lo que han surgido alternativas menos tediosas para lograr estos objetivos, como por ejemplo ctypes, CFFI, SWIG y Cython.
En este capítulo veremos en detalle esta última.
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
39.1. Cython: C Extensions for Python#
Recordemos que Python es un lenguaje interpretado con tipos dinámicos. Esto hace que cada operación tenga un overhead. Por ejemplo:
z = x + y
# overhead: Inferir el tipo de x
# overhead: Inferir el tipo de y
# Hacer la operación suma
# overhead: Darle el tipo adecuado a z
en cambio, Cython es un lenguaje de programación que le agrega a Python algunas propiedades de C y C++, una de ellas son los tipos estáticos, como muestra el siguiente ejemplo:
int x = 1
int y = 2
int z = x + y
# No hay que inferir el tipo de x, y, z
Esto hace que Cython sea menos flexible pero decenas de veces más rápido que Python. En términos de sintaxis Cython es casi tan simple como Python. Sin embargo a diferencia de Python, el lenguaje Cython debe compilarse.
El compilador de Cython convierte el código fuente en código C
Luego el código C se compila como un módulo de Python con la implementación CPython
Importante
Una vez compilado el código escrito en Cython este puede llamarse desde Python.
¿Por qué considerar Cython?
Cython es casi tan simple como Python y casi tan rápido como C
Con Cython se pueden llamar funciones y librerías de C
Cython se integra de buena manera con NumPy
Por ende Cython es muy atractivo para proyectos que usen Python y tengan requisitos de alto rendimiento. Estudiaremos la sintaxis de Cython mediante algunos ejemplos.
Ejemplo A lo largo de esta lección utilizaremos como ejemplo el cálculo de la Distancia euclidiana todos-con-todos
Sea un conjunto de con \(N\) datos con \(D\) atributos (matriz de NxD) donde queremos calcular la distancia euclidiana de cada dato con todos los demás, es decir una matriz donde el elemento \(ij\) es:
A continuación se muestran dos códigos que cumplen este propósito y obtienen un resultado equivalente:
El primero usa “Python puro” y calcula las distancias en un triple bucle
El segundo usa operaciones vectoriales de NumPy
Estudie ambos códigos y asegúrese de comprenderlos antes de continuar.
data = np.random.randn(1000, 2)
def distancia_pares(data):
N, D = data.shape
dist = np.zeros(shape=(N, N))
for i in range(N):
for j in range(i+1, N):
for k in range(D):
dist[i, j] += (data[i, k] - data[j, k])**2
dist[i, j] = np.sqrt(dist[i, j])
dist[j, i] = dist[i, j]
return dist
def distancia_pares_numpy(data):
return np.sqrt(np.sum((data.reshape(-1, 1, 2) - data.reshape(1, -1, 2))**2, axis=-1))
El resultado de ambas rutinas es equivalente:
np.allclose(distancia_pares(data), distancia_pares_numpy(data))
True
El tiempo que toma cada uno en completar es:
time_pure = %timeit -r3 -n1 -q -o distancia_pares(data)
time_pure.average
0.5916261283758407
time_numpy = %timeit -r10 -n5 -q -o distancia_pares_numpy(data)
time_numpy.average
0.013674410122912378
Por lo tanto, NumPy es:
time_pure.average/time_numpy.average
43.26520289050948
veces más rápido que Python puro
39.2. Cython desde IPython/Jupyter#
Instale cython en su ambiente de conda con:
conda install cython
Luego en IPython podemos cargar la extensión cython como se muestra a continuación.
%load_ext cython
con esto tendremos disponible la magia de bloque %%cython
Importante
Un bloque donde se use esta magia acepta lenguaje cython y se compila al ejecutarlo. Luego podremos llamar las funciones de ese bloque desde otros bloques regulares de Python.
Si surgen errores de compulación estos aparecen en la salida del bloque.
Nota
El bloque que empieza con %%cython está “desconectado” del resto del cuadernillo, por lo que debe tener sus propios import.
La magia tiene las siguientes opciones:
-a(annotate) retorna un profile linea a linea indicando con amarillo las llamadas a CPython (mientras más llamadas más lento es nuestro código)-+Usar C++ en lugar de C-cArgumentos de compilación-llibrerías para linkear a nuestro código-Ldirectorio con librerías-Idirectorio con cabeceras (include)
Veamos que ocurre al agregarle la magia al ejemplo anterior.
%%cython
import numpy as np
def distancia_pares_cython_ingenuo(data):
N, D = data.shape
dist = np.zeros(shape=(N, N))
for i in range(N):
for j in range(i+1, N):
for k in range(D):
dist[i, j] += (data[i, k] - data[j, k])**2
dist[i, j] = np.sqrt(dist[i, j])
dist[j, i] = dist[i, j]
return dist
np.allclose(distancia_pares_numpy(data), distancia_pares_cython_ingenuo(data))
True
time_cython_naive = %timeit -r3 -n1 -q -o distancia_pares_cython_ingenuo(data)
time_cython_naive.average
0.5202283986921733
time_pure.average/time_cython_naive.average
1.1372430452915634
time_numpy.average/time_cython_naive.average
0.026285397254915577
El resultado es idéntico pero como no hemos hecho ningún cambio, el tiempo de ejecución es sólo levemente mejor que la versión en Python puro.
A continuación veremos como “cythonizar” nuestro código para ganar rendimiento.
39.3. Mejora 1: Definiendo tipos en Cython#
En Cython se definen tipos estáticos con el keyword cdef seguido del tipo y luego el nombre. Por ejemplo una variable de tipo double llamada mi_variable sería:
cdef double mi_variable
Para los arreglos (ndarray) se usan “memory-views”. Por ejemplo declarar un memory-view para una arreglo de tres dimensiones sería:
cdef double [:, :, :] mi_arreglo
Y se puede ganar un poco más de rendimiento usando el operador ::1 para especificar si el arreglo es row-major (estilo C)
cdef double [:, :, ::1] mi_arreglo
o column-major (estilo Fortran)
cdef double [::1, :, :] mi_arreglo
Veamos como queda el ejemplo anterior introduciendo tipos estáticos.
%%cython
import numpy as np
def distancia_pares_cython_estatico(double [:, ::1] data):
# Definimos el tipo de N, D, dist y data
cdef int N = data.shape[0]
cdef int D = data.shape[1]
dist = np.zeros(shape=(N, N), dtype=np.double)
cdef double [:, ::1] dist_view = dist
# También definimos los índices, se puede usar int o Py_ssize_t
cdef int i, j, k
for i in range(N):
for j in range(i+1, N):
for k in range(D):
dist_view[i, j] += (data[i, k] - data[j, k])**2
dist_view[i, j] = np.sqrt(dist_view[i, j])
dist_view[j, i] = dist_view[i, j]
return dist
np.allclose(distancia_pares_numpy(data), distancia_pares_cython_estatico(data))
True
time_cython_static = %timeit -r10 -n5 -o -q distancia_pares_cython_estatico(data)
time_cython_static.average
0.14622879516333342
Speed-up con respecto a Python puro:
time_pure.average/time_cython_static.average
4.045893476144771
Speed-up con respecto a NumPy:
time_numpy.average/time_cython_static.average
0.09351379875378477
Con solo definir el tipo de data, dist, N, D y los índices hemos obtenido un speed-up importante con respecto a “Python puro”, aunque sigue siendo más lento que NumPy.
Si observaramos el código anotado por cython (compilado con -a) y revisamos la cantidad de llamadas a CPython de cada línea:
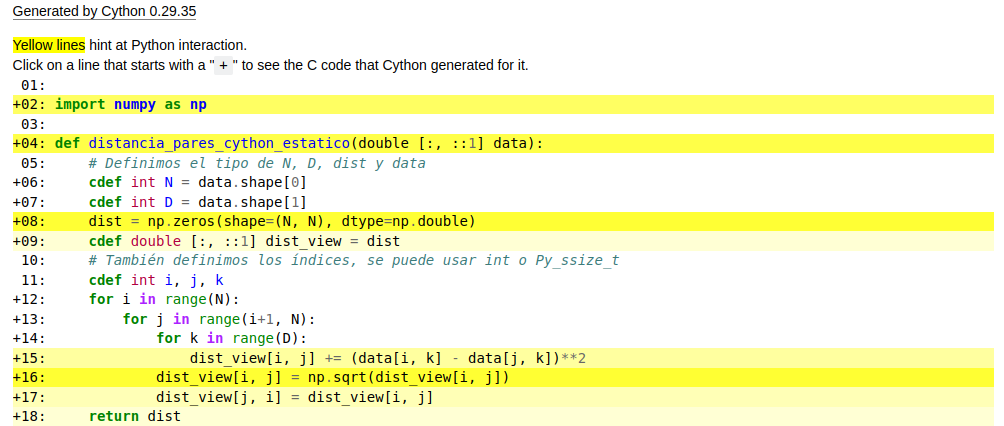
Podemos notar que la linea 16 es particularmente conflictiva:
involucra una gran cantidad de instrucciones
se llama NxN veces
esto se debe a que estamos usando la función de NumPy np.sqrt. Podemos obtener un rendimiento mucho mayor si usamos la implementación de raíz cuadrada de C.
39.4. Mejora 2: Llamando funciones de C desde Cython#
Es posible llamar funciones de C desde Cython de forma sencilla. Consideremos como ejemplo la función sqrt de la librería matemática estándar de C.
Para usar sqrt desde cython necesitamos:
Importar la función
sqrtdesde la cabeceramath.h. Esto se hace con el keywordcdef extern fromCompilar contra
libm
Veamos como queda nuestro ejemplo con esta modificación.
%%cython -l m
import numpy as np
cdef extern from "math.h":
double sqrt(double)
def distancia_pares_cython_sqrtC(double [:, ::1] data):
# Definimos el tipo de N, D, dist y data
cdef int N = data.shape[0]
cdef int D = data.shape[1]
dist = np.zeros(shape=(N, N), dtype=np.double)
cdef double [:, ::1] dist_view = dist
# También definimos los índices, se puede usar int o Py_ssize_t
cdef Py_ssize_t i, j, k
for i in range(N):
for j in range(i+1, N):
for k in range(D):
dist_view[i, j] += (data[i, k] - data[j, k])**2
dist_view[i, j] = sqrt(dist_view[i, j])
dist_view[j, i] = dist_view[i, j]
return dist
np.allclose(distancia_pares_numpy(data), distancia_pares_cython_sqrtC(data))
True
time_static_cfun = %timeit -r10 -n10 -q -o distancia_pares_cython_sqrtC(data)
time_static_cfun.average
0.001721905169542879
time_numpy.average/time_static_cfun.average
7.941442051970015
Con esta simple modificación hemos obtenido un tiempo de ejecución incluso menor que la implementación en NumPy.
Importante
Utilizando extern podemos hacer interfaces entre Python y casi cualquier código escrito en C.
39.5. Mejora 3: Deshabilitando comprobaciones para ir aun más rápido#
Podemos hacer nuestro código más rápido (y más inseguro) deshabilitando las verificaciones que Python realiza por defecto.
En Cython esto se logra usando decoradores que funcionan como directivas de compilación. Las opciones disponibles se encuentrán aquí. En este caso particular deshabilitaremos tres verificaciones: boundscheck y wraparound.
Al deshabilitarlas el código no comprobará si escribimos fuera del arreglo y tampoco traducirá índices negativos.
%%cython -l m
import cython
cimport numpy as npc
import numpy as np
# Por conveniencia podemos definir el tipo de data y dist con
cdef extern from "math.h":
double sqrt(double)
# Deshabilitamos las comprobaciones de Python:
@cython.boundscheck(False)
@cython.wraparound(False)
def distancia_pares_cython(double [:, ::1] data):
cdef int N = data.shape[0]
cdef int M = data.shape[1]
dist = np.zeros(shape=(N, N), dtype=np.double)
cdef double [:, ::1] dist_view = dist
cdef Py_ssize_t i, j, k
for i in range(N):
for j in range(i+1, N):
for k in range(M):
dist_view[i, j] += (data[i, k] - data[j, k])**2
dist_view[i, j] = sqrt(dist_view[i, j])
dist_view[j, i] = dist_view[i, j]
return dist
np.allclose(distancia_pares_numpy(data), distancia_pares_cython(data))
True
time_cython_static_cfun_nochecks = %timeit -r30 -n30 -q -o distancia_pares_cython(data)
time_cython_static_cfun_nochecks.average
0.0012969772641857466
time_numpy.average/time_cython_static_cfun_nochecks.average
10.543292084227312
Una modificación marginal en el código que aumenta el speed-up con respecto al caso anterior.
39.6. Mejora 4: Mayor flexibilidad con tipos de Cython fusionados#
Lo que hemos perdido al usar Cython con respecto a Python es la flexibilidad.
Si utilizamos un argumento que no sea double nuestra función en Cython retornará un error. Es nuestra responsabilidad evitar que esto ocurra.
data_float32 = data.astype(np.float32)
Si ejecutaramos la función distancia_pares_cython con datos de tipo float32 resultaría en una excepción.
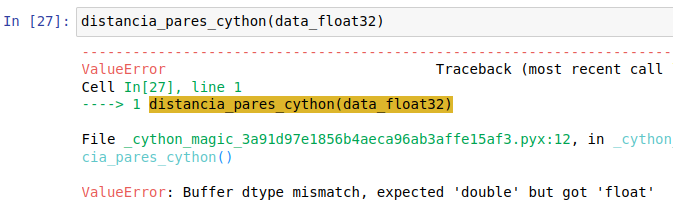
Podemos resolver este tipo de problemas con los “tipos fusionados” de Cython. Si queremos fusionar dos tipos de datos “tipo1” y “tipo2” bajo el nombre “tipo3”, lo declaramos con:
ctypedef fused tipo3:
tipo1
tipo2
Con esto Cython creará, de forma transparente, dos funciones en lugar de una. Cython ofrece algunos tipos fusionados listos para utilizar. Este es el caso del tipo fusionado floating, que combina números flotantes con precisión doble (float64) y simple (float32).
Modifiquemos la función distancia_pares_cython para que acepte los tipos double e float como un tipo fusionado. En este caso no debemos olvidar importar la definición de sqrtf de math.h.
%%cython -l m
import cython
from cython cimport floating
cimport numpy as npc
import numpy as np
cdef extern from "math.h":
float sqrtf(float) #Definición para float32
double sqrt(double) # Definición para float64
@cython.boundscheck(False)
@cython.wraparound(False)
def distancia_pares_cython_multitipo(floating [:, ::1] data):
cdef int N = data.shape[0]
cdef int M = data.shape[1]
# Comprobamos el tipo antes de crear el arreglo de numpy
if floating is double:
dist = np.zeros(shape=(N, N), dtype=np.float64)
else:
dist = np.zeros(shape=(N, N), dtype=np.float32)
cdef floating [:, ::1] dist_view = dist
cdef Py_ssize_t i, j, k
for i in range(N):
for j in range(i+1, N):
for k in range(M):
dist_view[i, j] += (data[i, k] - data[j, k])**2
if floating is double:
dist_view[i, j] = sqrt(dist_view[i, j])
else:
dist_view[i, j] = sqrtf(dist_view[i, j])
dist_view[j, i] = dist_view[i, j]
return dist
Los resultados son equivalentes a NumPy tanto si se usa precisión doble o simple:
np.allclose(distancia_pares_numpy(data),
distancia_pares_cython_multitipo(data))
True
np.allclose(distancia_pares_numpy(data_float32),
distancia_pares_cython_multitipo(data_float32))
True
time_numpy_f32 = %timeit -r10 -n10 -q -o distancia_pares_numpy(data_float32)
time_numpy_f32.average
0.012807978300843386
time_cython_f32 = %timeit -r30 -n30 -q -o distancia_pares_cython_multitipo(data_float32)
time_cython_f32.average
0.0005656014873077058
time_numpy_f32.average/time_cython_f32.average
22.644880871530354
El speed-up con double es practicamente equivalente al caso anterior.
time_cython = %timeit -r30 -n30 -q -o distancia_pares_cython_multitipo(data)
time_numpy.average/time_cython.average
10.50371420453281
39.7. Usando Cython fuera de jupyter#
Ver también
Lecturas sobre temas específicos de Cython: