13. Entrenamiento de redes neuronales con PyTorch#
13.1. Construcción de redes neuronales#
PyTorch nos ofrece la clase tensor y las funcionalidades de autograd. Estas poderosas herramientas nos dan todo lo necesario para construir y entrenar redes neuronales artificiales.
Para facilitar aun más estas tareas PyTorch tiene módulos de alto nivel que implementan:
Modelo base de red neuronal:
torch.nn.ModuleDistintos tipos de capas, funciones de activación y funciones de costo:
torch.nnDistintos algoritmos de optimización basados en gradiente descedente:
torch.optim
Una red neuronal en PyTorch es una clase de Python que hereda de torch.nn.Module. Como mínimo esta clase debe implementar las funciones __init__ y forward
El constructor define las capas que se utilizaran
Heredar de
nn.Modulehace que los parámetros de las capas queden registrados por la máquina de estado de PyTorchLa función
forwardrecibe como argumento los datos de entrada y retorna la predicción del modelo, es decir que define como se conectan las capas
Nota
La función forward() actua como la función __call__() de Python, es decir que se creamos una objeto model que herada de nn.Module llamar model.forward(x) es equivalente a model(x)
13.1.1. Capa completamente conectada#
Una capa completamente conectada (fully-connected) también llamada capa densa, implementa la siguiente operación:
donde \(x\) son los datos que entran en la capa y \(w,b\) son los parámetros (pesos y sesgos) de la capa.
Esta capa está implementada en Pytorch como torch.nn.Linear. El constructor de este objeto espera la dimensionalidad (número de neuronas) de entrada y salida de la capa. Por ejemplo para crear una capa con tres entradas y dos salidas utilizaríamos:
import torch
import torch.nn as nn
dense = torch.nn.Linear(3, 2)
dense.weight, dense.bias
(Parameter containing:
tensor([[ 0.1703, -0.4679, -0.2857],
[ 0.0995, -0.0204, -0.0667]], requires_grad=True),
Parameter containing:
tensor([ 0.4130, -0.4899], requires_grad=True))
Una vez creada la podemos evaluar con dense(data) o dense.forward(data):
dense(torch.Tensor([0, 0, 0]))
tensor([ 0.4130, -0.4899], grad_fn=<AddBackward0>)
Nota
Las capas son a su vez instancias de torch.nn.Module. Es decir que un módulo puede tener otros módulos anidados.
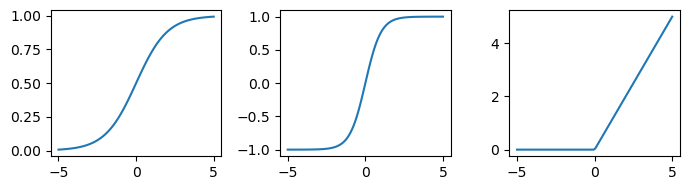
13.1.2. Funciones de activación#
Las funciones de activación más comunes de la literatura están implementadas como clases en torch.nn
Veamos algunos ejemplos para aprender a utilizarlas:
%matplotlib inline
from matplotlib import pyplot as plt
data = torch.linspace(-5, 5, steps=100)
activations = [nn.Sigmoid(), nn.Tanh(), nn.ReLU()]
fig, ax = plt.subplots(1, len(activations), figsize=(7, 2), tight_layout=True)
for activation, ax_ in zip(activations, ax):
ax_.plot(data, activation(data))
13.1.3. Perceptrón multicapa (MLP) en Pytorch#
Considerando lo anteriormente mencionado la implementación de un perceptrón multicapa (multilayer perceptron) con una capa oculta y función de activación sigmoide en PyTorch sería:
import torch
import torch.nn as nn
class MultiLayerPerceptron(nn.Module):
def __init__(self, input_dim, hidden_dim, output_dim):
super(type(self), self).__init__()
self.hidden = nn.Linear(input_dim, hidden_dim)
self.output = nn.Linear(hidden_dim, output_dim)
self.activation = nn.Sigmoid()
def forward(self, x):
x = self.activation(self.hidden(x))
return self.output(x)
Crear una capa Linear hace que se registren sus parámetros weight y bias en el grafo. Inicialmente los parámetros tienen valores aleatorios
model = MultiLayerPerceptron(input_dim=2, output_dim=1, hidden_dim=2)
model.hidden.weight, model.hidden.bias, model.output.weight, model.output.bias
(Parameter containing:
tensor([[-0.3532, -0.6750],
[ 0.2961, -0.6165]], requires_grad=True),
Parameter containing:
tensor([-0.6416, -0.3000], requires_grad=True),
Parameter containing:
tensor([[-0.5612, -0.6022]], requires_grad=True),
Parameter containing:
tensor([-0.3252], requires_grad=True))
PyTorch también admite una forma “más funcional” de crear modelos utilizando torch.nn.Sequential.
El modelo anterior sería:
model = nn.Sequential(nn.Linear(2, 2),
nn.Sigmoid(),
nn.Linear(2, 1))
Esta forma de crear modelos es más concisa pero menos reutilizable.
13.2. Funciones de costo y optimizadores#
Para entrenar una red neuronal debemos definir
Una función de costo: Aquello que vamos a minimizar
Un algoritmo de optimización: De que forma vamos a minimizar
Las funciones de costo típicas están implementadas como clases en el módulo torch.nn. Por ejemplo la clase MSELoss representa el error cuadrático medio, que se define como:
donde \(y\) es la etiqueta (vector con D dimensiones) e \(\hat y\) es la predicción del modelo.
Nota
El MSE se utiliza en problemas de regresión (etiqueta con valores reales). Cuando entrenamos con esta función de costo debemos asegurarnos de que el modelo tenga tantas unidades de salida como dimensiones tenga la etiqueta.
La clase CrossEntropyLoss representa la entropía cruzada, que se define como:
donde \(y_c \in \{0,1\}\), \(\hat y_c \in [0,1]\) y \(\sum_{c=1}^C \hat y_c = 1\).
Nota
Esta función de costo se utiliza en problemas de clasificación de \(C\) clases. Cuando entrenamos con esta función de costo debemos asegurarnos de que el modelo tenga tantas unidades de salida como clases tenga el problema.
Advertencia
La implementación de entropía cruzada de torch espera que las etiquetas \(y\) estén en formato categórico, es decir como un entero con valor \(0, 1, 2, \ldots, C-1\). Adicionalmente espera que las predicciones \(\hat y\) estén en formato logits (números reales pre-activados).
Luego de crear una instancia de estas clases las podemos llamar cpmo una función. Por ejemplo si queremos calcular el error medio cuadrático entre dos tensores:
criterion = torch.nn.MSELoss(reduction='mean')
loss = criterion(output, target)
Nota
También podemos crear nuestra propia función de costo. Por ejemplo podríamos lograr lo mismo que MSELoss habiendo definido criterion = lambda ypred, y : (y-ypred).pow(2).mean()
Los algoritmos de optimización están implementados en el módulo torch.optim. Hoy en día los más utilizados son:
Optimizador |
Descripción |
|---|---|
Gradiente descedente estocástico con momentum |
|
Gradiente descedente con tasa de aprendizaje adaptiva |
Para crear un objeto optimizador debemos entregar como argumento los parámetros del modelo y las argumentos específicos del optimizador. Por ejemplo para SGD:
optimizer = torch.optim.SGD(model.parameters(), lr=1e-3, momentum=0, weight_decay=0)
donde:
lrEs la tasa de aprendizaje. Debe ser un valor pequeño para no desestabilizar el entrenamiento, pero no tan pequeño para enlentencerlo demasiadomomentumes la tasa de momentum. Podemos utilizar un valor mayor que cero para evitar estancamiento en mínimos localesweight_decaycontrola la regularización (norma L2) de los parámetros. Podemos utilizar un valor mayor que cero para evitar sobreajuste
Una vez creado podemos llamar su función principal step() la cual realiza una actualización de parámetros de acuerdo a las derivadas calculadas con loss.backward().
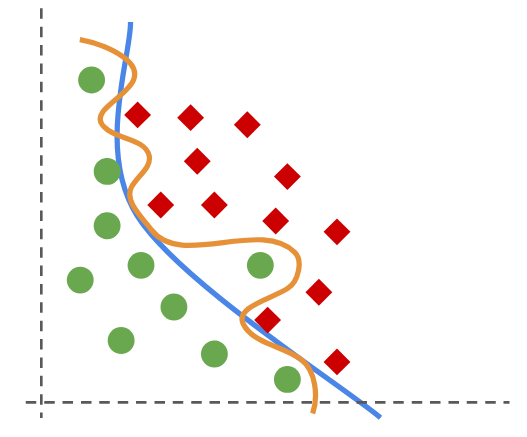
13.3. Ajustando un modelo paso a paso#
Consideremos el siguiente dataset \(X\) con etiqueta \(Y\). El dataset tiene cuatro ejemplos separados en dos clases. Los ejemplos tienen cada uno dos atributos.
X = torch.tensor([[-1.0, -1.0],
[-1.0, 1.0],
[1.0, -1.0],
[1.0, 1.0]])
Y = torch.tensor([0, 0, 0, 1])
n_features, n_classes = X.shape[1], len(Y.unique())
Utilizaremos CrossEntropyLoss para entrenar por lo tanto consideramos dos unidades de salida en la última capa.
n_hidden = 3
model = nn.Sequential(nn.Linear(n_features, n_hidden),
nn.Sigmoid(),
nn.Linear(n_hidden, n_classes))
criterion = torch.nn.CrossEntropyLoss(reduction='sum')
model
Sequential(
(0): Linear(in_features=2, out_features=3, bias=True)
(1): Sigmoid()
(2): Linear(in_features=3, out_features=2, bias=True)
)
Como optimizador utilizaremos Adam:
optimizer = torch.optim.Adam(model.parameters(), lr=1e-2)
Primero realizamos una predicción (inferencia) con:
hatY = model(X)
hatY
tensor([[-0.0495, 0.5699],
[ 0.1917, 0.6147],
[-0.0600, 0.4073],
[ 0.1669, 0.4399]], grad_fn=<AddmmBackward0>)
Luego calculamos la función de pérdida:
loss = criterion(hatY, Y)
loss
tensor(3.4967, grad_fn=<NllLossBackward0>)
En seguida calculamos el gradiente de la función de pérdida:
loss.backward()
Los gradientes se guardan en el atributo grad de todos los pesos y sesgos (parámetros) de la red:
model[0].weight.grad, model[0].bias.grad
(tensor([[0.0197, 0.0147],
[0.1103, 0.1128],
[0.0111, 0.0088]]),
tensor([-0.0283, -0.1992, -0.0116]))
Finalmente actualizamos los parámetros usando la función step de nuestro optimizador
Los parámetros actuales de la primera capa son:
model[0].weight, model[0].bias
(Parameter containing:
tensor([[ 0.7040, 0.2434],
[ 0.5366, 0.6847],
[-0.6445, 0.3375]], requires_grad=True),
Parameter containing:
tensor([ 0.5884, -0.0226, -0.2966], requires_grad=True))
optimizer.step()
Los parámetros luego de hacer la actualización son:
model[0].weight, model[0].bias
(Parameter containing:
tensor([[ 0.6940, 0.2334],
[ 0.5266, 0.6747],
[-0.6545, 0.3275]], requires_grad=True),
Parameter containing:
tensor([ 0.5984, -0.0126, -0.2866], requires_grad=True))
Truco
Para interpretar la salida de la red como probabilidades podemos aplicar una activación nn.Softmax(dim=1) como se muestra a continuación.
nn.Softmax(dim=1)(hatY).detach()
tensor([[0.3499, 0.6501],
[0.3958, 0.6042],
[0.3853, 0.6147],
[0.4322, 0.5678]])
Nota
Cada columna representa a una clase y cada fila a un ejemplo. Los valores están siempre entre cero y uno, y además las filas suman uno.
En general el proceso de ajuste se realiza iterativamente durante un cierto número de pasos o “épocas” de entrenamiento, tal como se muestra a continuación:
for nepoch in range(1000):
hatY = model.forward(X)
optimizer.zero_grad()
loss = criterion(hatY, Y)
loss.backward()
optimizer.step()
if nepoch % 100 == 0:
print(f"{nepoch} {loss.item():0.2f} {torch.nn.Softmax(dim=1)(hatY)[:, 1].detach()}")
0 3.43 tensor([0.6393, 0.5906, 0.6028, 0.5537])
100 1.38 tensor([0.0801, 0.1442, 0.2093, 0.4026])
200 0.52 tensor([0.0110, 0.0650, 0.0681, 0.6931])
300 0.22 tensor([0.0031, 0.0271, 0.0289, 0.8481])
400 0.12 tensor([0.0014, 0.0140, 0.0156, 0.9118])
500 0.08 tensor([8.5396e-04, 8.5738e-03, 9.7344e-03, 9.4210e-01])
600 0.06 tensor([5.7724e-04, 5.8299e-03, 6.6357e-03, 9.5879e-01])
700 0.04 tensor([4.2245e-04, 4.2423e-03, 4.8179e-03, 9.6900e-01])
800 0.03 tensor([3.2588e-04, 3.2357e-03, 3.6612e-03, 9.7575e-01])
900 0.03 tensor([2.6089e-04, 2.5543e-03, 2.8788e-03, 9.8046e-01])
Nota
Paso a paso, el error (función de costo) disminuye y la predicción del modelo se acerca a la etiqueta
13.4. Entrenamiento por minibatches#
Si el dataset del problema es de gran tamaño no es conveniente utilizar el conjunto completo para calcular la función de costo y derivar. En general actualizamos el modelo presentándole subconjuntos (minibatches) del dataset.
Nota
Esto es lo que se conoce como la estimación estocástica del gradiente descedente (stochastic gradient descent, SGD).
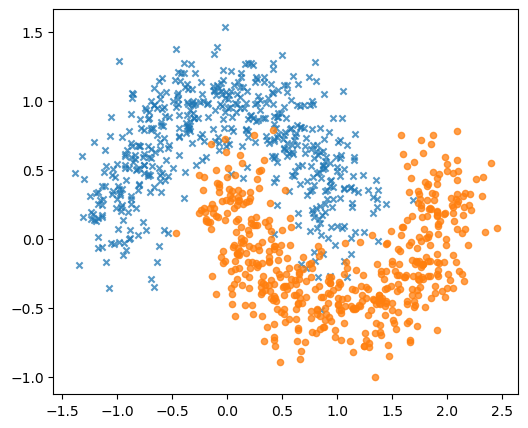
A continuación veremos algunas clases de PyTorch que facilitan la interacción entre el modelo y el conjunto de entrenamiento. A modo de ejemplo crearemos un conjunto de datos sintético con dos atributos, dos clases y que no es linealmente separable:
import numpy as np
import sklearn.datasets
np.random.seed(0)
X, y = sklearn.datasets.make_moons(n_samples=1000, noise=0.2)
fig, ax = plt.subplots(figsize=(6, 5))
for k, marker in enumerate(['x', 'o']):
mask = y == k
ax.scatter(X[mask, 0], X[mask, 1], s=20, marker=marker, alpha=0.75)
El súbmodulo torch.utils.data tiene los objetos que facilitan la interacción con datos. Sus clases principales son:
Dataset: Clase abstracta que representa a un conjunto de datosSubset: Clase que representa una partición de un conjunto de datos, por ejemplo la partición de entrenamiento o validaciónDataLoader: Generador que recibe un conjunto de datos y retorna subconjuntos (minibatches) iterativamente
El primer paso es crear una clase que represente nuestro conjunto de datos en particular, esta clase debe heredar de Dataset y debe implementar las funciones __len__ y __getitem__:
import torch.utils.data as data
class MyDataset(data.Dataset):
def __init__(self, data: np.ndarray, labels: np.ndarray):
self.data = torch.from_numpy(data.astype('float32'))
self.labels = torch.from_numpy(labels.astype('int'))
def __getitem__(self, idx: int):
return (self.data[idx], self.labels[idx])
def __len__(self):
return len(self.labels)
dataset = MyDataset(X, y)
dataset[0], len(dataset)
((tensor([2.0861, 0.5740]), tensor(1)), 1000)
donde
El constructor recibe los datos y los convierte en formato tensor
__getitem__, recibe un índice y retorna una tupla (dato, etiqueta)__len__retorna la cantidad de ejemplos del conjunto de datos
Para crear particiones de este dataset podemos utilizar la función random_split del módulo data. Esta función recibe el dataset, los tamaños de cada partición y una semilla aleatoria:
train_set, valid_set, test_set = data.random_split(dataset, [600, 200, 200],
generator=torch.Generator().manual_seed(1234))
Finalmente creamos objetos DataLoader para obtener minibatches de entrenamiento, validación y prueba con:
train_loader = data.DataLoader(train_set, shuffle=True, batch_size=32)
valid_loader = data.DataLoader(valid_set, shuffle=False, batch_size=128)
test_loader = data.DataLoader(test_set, shuffle=False, batch_size=128)
Los argumentos principales de esta clase son:
Un objeto
DatasetoSubsetshuffle: Booleano que índica si los ejemplos se muestran de forma ordenada o desordenadabatch_size: Entero que indica la cantidad de ejemplos por minibatch
Importante
Es conveniente presentar el conjunto de entrenamiento en distinto orden en cada época. Utilice shuffle=True para este conjunto.
Una vez creado el objeto DataLoader se puede ocupar de forma equivalente a otros iteradores de Python:
for batchx, batchy in train_loader:
break
batchx.T, batchy
(tensor([[ 0.2742, 0.0838, 0.3535, 1.0220, 0.5325, 0.9625, -0.1301, 0.7547,
0.4513, 0.4570, -0.7164, -0.3492, 0.9425, 0.7684, 0.0605, -0.3334,
0.0351, 0.2826, -0.1815, 0.4193, 1.3026, -0.3905, -1.2129, 0.4229,
0.8711, -0.9832, -0.2609, -0.3761, -0.2786, 0.2538, 0.7121, -0.7011],
[ 1.1606, 0.0487, 0.7483, 0.5886, -0.3438, 0.2852, 0.1867, 0.3459,
0.2463, -0.5059, 0.8689, 0.7954, 0.4273, -0.6735, -0.0425, 0.9329,
-0.2298, -0.0536, 0.9742, -0.4464, -0.5140, 0.2952, 0.6996, 0.8891,
0.5274, -0.0205, 0.8748, 0.8369, 0.5381, 0.7022, 0.6715, 0.4590]]),
tensor([0, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, 0, 0, 1, 1, 0, 1, 1, 0, 1, 1, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0]))
13.5. Esquema general de entrenamiento en PyTorch#
Habiendo definido el modelo, el criterio, el optimizador y los datos, el esquema general de entrenamiento de un modelo en PyTorch sería:
for epoch in range(num_epochs): # Durante un cierto número de épocas
for minibatch in data: # Para cada minibatch de datos
optimizer.zero_grad() # Limpiamos los gradientes
x, y = minibatch # Desempaquetamos
yhat = model.forward(x) # Predecimos
loss = criterion(yhat, y) # Evaluamos
loss.backward() # Calculamos los gradientes
optimizer.step() # Actualizamos los parámetros
donde
Una época es una presentación completa del conjunto de entrenamiento
Un minibatch es un subconjunto del conjunto de entrenamiento
Adicionalmente, debemos considerar un bucle de validación donde sólo realizamos predicción y evaluación de la loss con el objetivo de detectar sobre-ajuste
- Early Stopping
Detención del entrenamiento cuando la loss de validación no haya disminuido durante una cierta cantidad de épocas (paciencia). Se utiliza para evitar el sobreajuste
Importante
En cada época, verifique si el error de validación ha alcanzado un nuevo mínimo. Si es así guarde un respaldo del modelo utilizado torch.save.
A continuaciones se ponen en práctica estos criterios utilizando PyTorch. Por conveniencia se implementan las siguientes funciones:
update_step: Recibe un minibatch y actualiza los parámetrosevaluation_step: Recibe un minibtach y evalúa el criterio de optimizacióntrain_one_epoch: Realiza una época de entrenamiento. Se encarga de guardar el modelo si es el mejor encontrado hasta ese punto
def update_step(data, label):
prediction = model(data)
optimizer.zero_grad()
loss = criterion(prediction, label)
loss.backward()
optimizer.step()
return loss.item()
def evaluate_step(data, label):
prediction = model(data)
loss = criterion(prediction, label)
return loss.item()
def train_one_epoch(epoch):
train_loss, valid_loss = 0.0, 0.0
for batchx, batchy in train_loader:
train_loss += update_step(batchx, batchy)
for batchx, batchy in valid_loader:
valid_loss += evaluate_step(batchx, batchy)
# Guardar modelo si es el mejor hasta ahora
global best_valid_loss
if epoch % 10 == 0:
if valid_loss < best_valid_loss:
best_valid_loss = valid_loss
torch.save({'epoca': epoch,
'model_state_dict': model.state_dict(),
'optimizer_state_dict': optimizer.state_dict(),
'loss': valid_loss},
'best_model.pt')
return train_loss/len(train_loader.dataset), valid_loss/len(valid_loader.dataset)
Nota
Para entrenar en GPU sería necesario
que el modelo esté en memoría de GPU
enviar los datos y etiquetas a GPU antes de realizar los cálculos.
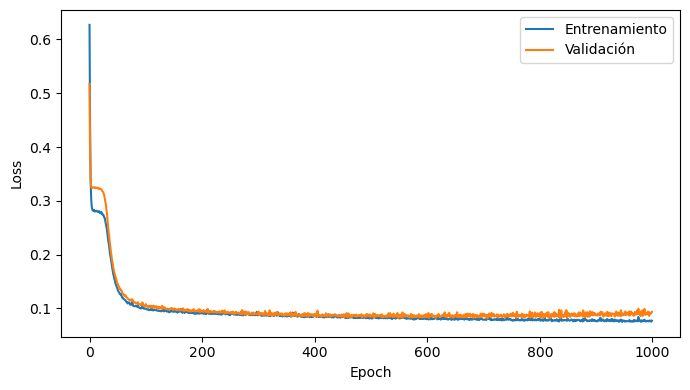
En este caso el modelo se entrena por 500 épocas
%%time
def my_model(n_hidden: int):
return nn.Sequential(nn.Linear(2, n_hidden), nn.Tanh(), nn.Linear(n_hidden, 2))
model = my_model(5)
criterion = torch.nn.CrossEntropyLoss(reduction='sum')
optimizer = torch.optim.Adam(model.parameters(), lr=1e-2)
max_epochs, best_valid_loss = 1000, np.inf
running_loss = np.zeros(shape=(max_epochs, 2))
for epoch in range(max_epochs):
running_loss[epoch] = train_one_epoch(epoch)
fig, ax = plt.subplots(figsize=(7, 4), tight_layout=True)
ax.plot(running_loss[:, 0], label='Entrenamiento')
ax.plot(running_loss[:, 1], label='Validación')
ax.set_xlabel('Epoch')
ax.set_ylabel('Loss')
ax.legend();
CPU times: user 4min 3s, sys: 362 ms, total: 4min 3s
Wall time: 1min 1s
Cargamos la información serializada del modelo utilizado torch.load
saved_model = torch.load('best_model.pt')
El mejor modelo se obtuvo en la época
saved_model['epoca']
520
Podemos asignar los mejores parámetros al modelo anterior con:
model = my_model(5)
model.load_state_dict(saved_model['model_state_dict'])
<All keys matched successfully>
Podemos evaluar el mejor modelo en el conjunto de test para analizar su capacidad de generalización
from sklearn.metrics import classification_report
ytrue, ypred = [], []
for x, y_ in test_loader:
ypred.append(nn.Softmax(dim=1)(model(x)).detach().argmax(dim=1))
ytrue.append(y_)
ytrue, ypred = np.concatenate(ytrue), np.concatenate(ypred)
print(classification_report(ytrue, ypred))
precision recall f1-score support
0 0.96 0.96 0.96 104
1 0.96 0.96 0.96 96
accuracy 0.96 200
macro avg 0.96 0.96 0.96 200
weighted avg 0.96 0.96 0.96 200
En este caso también podemos visualizar el resultado de predicción en el espacio de características:
x1_test = np.arange(-2, 3, 0.05, dtype=np.float32)
x2_test = np.arange(-1.5, 2, 0.05, dtype=np.float32)
xx1_test, xx2_test = np.meshgrid(x1_test, x2_test)
x_test = torch.from_numpy(np.stack((xx1_test.ravel(), xx2_test.ravel())).T)
prob_test = nn.Softmax(dim=1)(model(x_test)).detach()[:, 1].reshape(xx1_test.shape)
fig, ax = plt.subplots(figsize=(6, 5))
ax.pcolormesh(x1_test, x2_test, prob_test, cmap=plt.cm.RdBu_r)
for k, marker in enumerate(['x', 'o']):
mask = y == k
ax.scatter(X[mask, 0], X[mask, 1], c='k', s=20, marker=marker, alpha=0.5)
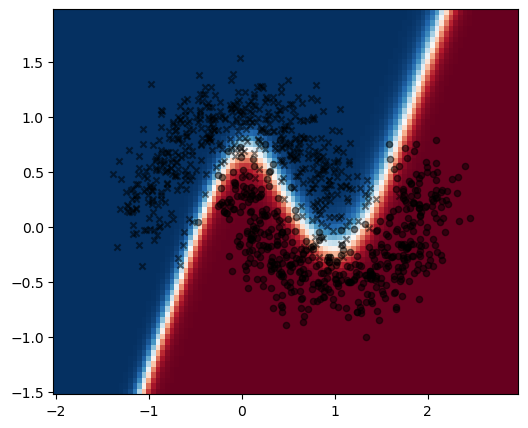
El resultado en la capa de salida tiene forma no lineal. Sin embargo, si inspeccionemos la salida de la capa oculta, que en este caso tiene 5 neuronas:
fig, ax = plt.subplots(1, 5, figsize=(10, 2), tight_layout=True)
for ax_, layer, w in zip(ax.ravel(), model[0](x_test).T, model[2].weight.detach()[0]):
ax_.pcolormesh(xx1_test, xx2_test,
nn.Tanh()(layer).reshape(xx1_test.shape).detach(),
cmap=plt.cm.RdBu_r)
ax_.set_title(f"{w.item():0.4f}")
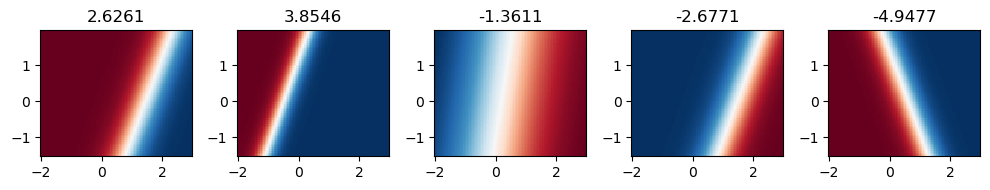
Nota
La capa de salida combina los hiperplanos de la capa oculta. Sobre cada neurona oculta se muestra su peso en la capa de salida. Mientras más cercano a cero, menos relevante es su aporte.
13.6. Diagnósticos a partir de curvas de aprendizaje#
Podemos diagnosticar el entrenamiento observando la evolución de la función de costo .
Importante
Siempre visualice la loss en ambos conjuntos: entrenamiento y validación.
Veamos algunos casos.
Ambas curvas en descenso
Si las curvas se ven así:
epochs = np.arange(1, 500)
loss_train = (epochs)**(-1/10) + 0.01*np.random.randn(len(epochs))
loss_valid = (epochs)**(-1/10) + 0.01*np.random.randn(len(epochs)) + 0.1
fig, ax = plt.subplots(figsize=(6, 3))
ax.plot(epochs, loss_train, lw=2, label='entrenamiento')
ax.plot(epochs, loss_valid, lw=2, label='validación')
ax.set_ylim([0.5, 1.05])
plt.legend();
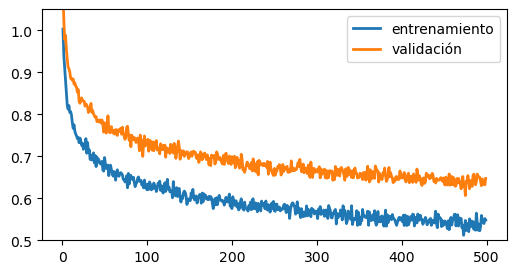
En este caso deberías continuar con el entrenamiento, pues no hay señal de convergencia
Sobreajuste temprano
Si las curvas se ven así:
epochs = np.arange(1, 500)
loss_train = (epochs)**(-1/10) + 0.01*np.random.randn(len(epochs))
loss_valid = (epochs)**(-1/10) + 0.00001*(epochs)**2 +0.01*np.random.randn(len(epochs)) + 0.01
fig, ax = plt.subplots(figsize=(6, 3))
ax.plot(epochs, loss_train, lw=2, label='entrenamiento')
ax.plot(epochs, loss_valid, lw=2, label='validación')
ax.set_ylim([0.5, 1.05])
plt.legend();
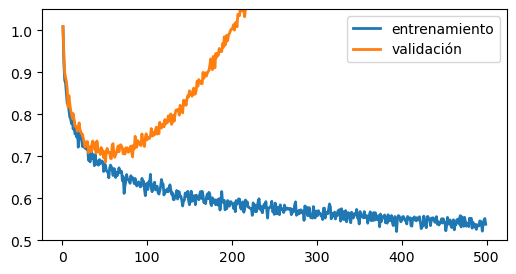
Significa que tu modelo se ha sobreajustado. Considere las siguientes opciones:
Implementar un modelo más sencillo, por ejemplo disminuyendo la cantidad de neuronas en la capa oculta.
Conseguir más datos de entrenamiento
Incorporar regularización, por ejemplo weight decay
Error en el código o mal punto de partida
Si tus curvas se ven así:
epochs = np.arange(1, 500)
loss_train = 1.0 + 0.01*np.random.randn(len(epochs))
loss_valid = 1.0 + 0.01*np.random.randn(len(epochs)) + 0.01
fig, ax = plt.subplots(figsize=(6, 3))
ax.plot(epochs, loss_train, lw=2, label='entrenamiento')
ax.plot(epochs, loss_valid, lw=2, label='validación')
#ax.set_ylim([0.5, 1.05])
plt.legend();
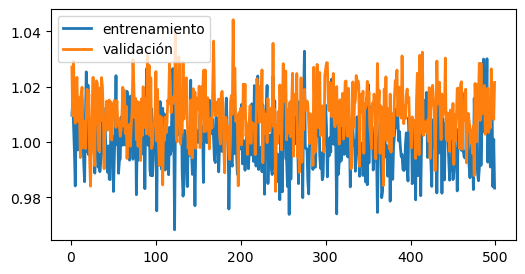
Revice que su código no tenga bugs. Algunas causas comunes de error son:
el modelo no esté recibiendo adecuadamente los datos y/o la etiqueta
los datos tiene un rango muy grande y los gradientes explotan: normalice o reescale los datos
la función de costo no es la adecuada para el problema
la tasa de aprendizaje es demasiado alta
También puede deberse a una muy mala inicialización aleatoria, reinicia el entrenamiento para comprobar si es el caso.